Der Nobelpreis für Chemie 2024 würdigt die Arbeit von David Baker, Professor an der University of Washington in Seattle, sowie die von Demis Hassabis und John Jumper von DeepMind – einem Google-Unternehmen, das in der Öffentlichkeit für die Entwicklung künstlicher Computersoftware bekannt ist Intelligenz AlphaGo (das den Go-Weltmeister 2017 besiegte) und dann AlphaFold.
Durch die Arbeit an Proteinen mithilfe von Computerwerkzeugen haben diese drei Forscher zu einem besseren Verständnis dieser lebenswichtigen Moleküle beigetragen.
Der Nobelpreis für Chemie 2024 würdigt zwei spektakuläre Fortschritte in der Wissenschaft der Proteine – Moleküle, die eine entscheidende Rolle in zellulären Prozessen spielen. Daher ist es wichtig zu verstehen, wie Proteine interagieren, um die Mechanismen aufzudecken, die das Leben steuern, und um künftig Aufschluss über die Anfälligkeit und Schwere von Krankheiten zu geben, während gleichzeitig die Entschlüsselung der Zusammenhänge zwischen Genen, Krankheiten und Medikamenten erleichtert wird.
Proteine sind Ketten aus Aminosäuren, die sich ineinander falten. Bemerkenswert ist, dass die dreidimensionale Form in der Aminosäuresequenz kodiert ist. Das bedeutet, dass wir diese dreidimensionale Struktur allein aus der Aminosäuresequenz vorhersagen können sollten. Das ist der springende Punkt Vorhersage der Proteinstruktur.
Andererseits können wir uns, sobald die Struktur bekannt ist, fragen, ob wir eine Sequenz finden können, die sie erzeugt: Dies ist, was wir nennen Konzeption oder Design von Proteinenalso die Entstehung völlig neuer Proteine, die es in der Natur noch nie gab.
Dieser Nobelpreis unterstreicht die Leistungsfähigkeit der Computerbiologie und die immensen Möglichkeiten, die sie den Biowissenschaften in den kommenden Jahren eröffnet.
David Bakers Beitrag: die Entwicklung von Proteinen, die es in der Natur noch nie gegeben hat
Das Problem des Proteindesigns wurde 2003 von David Baker gelöst, wobei die Entstehung von Proteinen noch nie zuvor beobachtet wurde. Mit der Software Rosetta können Sie eine Sequenz entwerfen, die einer vorgegebenen Struktur entspricht.
Seitdem hat David Baker eine neue Welt beispielloser Proteinstrukturen eröffnet. Für ihn bestand das Ziel darin, zu verstehen, wie man Proteinstrukturen durch die Gestaltung neuer und komplexer Proteine „beherrscht“, um die Natur für unsere eigenen Zwecke zu nutzen.
PS Huang, SE Boyken und D. Baker, Nature, 537, 320–327, 2016
Durch die Kombination von kombinatorischer Optimierung, Chemie und Physik ermöglicht Rosetta Wissenschaftlern nun die Entwicklung von Proteinen mit spektakulären Formen und Funktionen, die bisher unbekannt waren.
Der Beitrag von Demis Hassabis und John Jumper: Vorhersage der Struktur von Proteinen anhand der Aminosäuresequenz
Demis Hassabis und John Jumper versuchten, eine seit Jahrzehnten offene Frage zu klären: die Rekonstruktion einer Proteinstruktur aus ihrer Aminosäuresequenz. Es gelang ihnen, den Proteincode zu entschlüsseln und so die Struktur fast aller nicht ungeordneten natürlichen Proteine vorherzusagen.
Im Jahr 2020 präsentierten sie AlphaFold2, das im Vergleich zu bekannten kristallographischen Strukturen eine Genauigkeit von etwa 90 % erreichte, was mit experimentellen Fehlern bei der Strukturbestimmung übereinstimmt.
Weiterlesen: Künstliche Intelligenz zur Herausforderung des Proteindesigns: die Erfolge und Grenzen von AlphaFold
Dieser spektakuläre Fortschritt erschütterte die Welt der Biologie: Über Nacht wuchs die den Forschern zugängliche „Proteinbibliothek“ von 200.000 kristallografischen Strukturen auf 200 Millionen AlphaFold2-Modelle, die fast identisch mit den Strukturen von Kristallen sind!
Jetzt kann ein Biologe, Virologe oder Arzt eine Aminosäuresequenz an AlphaFold2 übermitteln und sofort deren dreidimensionale Struktur erhalten. Diese präzise Visualisierung der Position von Atomen ermöglicht die Analyse der Schwachstellen eines Proteins während seiner Wechselwirkungen, was zur Entwicklung von Medikamenten genutzt werden kann, die diese Wechselwirkungen blockieren. Es ermöglicht auch die Identifizierung von Rückständen, die gegenüber zukünftigen Mutationen empfindlich sind, sowie von Rückständen, die an Interaktionen mit potenziellen molekularen Partnern beteiligt sind.
Dies eröffnet den Weg zur Entdeckung neuer, oft unerwarteter Aspekte der Biologie eines Organismus sowie der Interaktionen zwischen verschiedenen Organismen.
Ein Chemie-Nobelpreis an der Schnittstelle von Biologie und Informatik
Diese beiden grundlegenden Fortschritte für Chemie und Biologie wären ohne die wesentliche Kombination mehrerer Elemente nicht möglich gewesen: einerseits die Fülle an Proteinsequenzen, die in unseren Datenbanken verfügbar sind, und die große Anzahl von Strukturen, die dank der Arbeit von Experimentalbiologen kristallisiert wurden, und so weiter Zum anderen dank der Fortschritte in der künstlichen Intelligenz, die es ermöglicht, riesige Datenmengen zu erforschen und auf der Grundlage von Deep Learning und GPU-Technologie komplexe, hochpräzise Informationen abzuleiten.
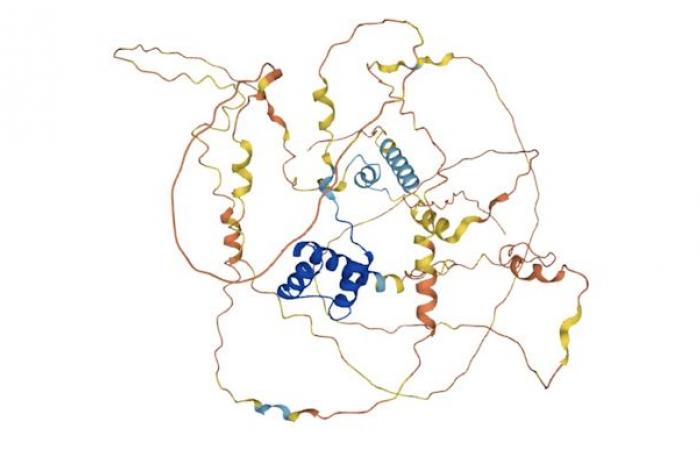
ARX-Protein, AlphaFold-Datenbank. Mutationen in diesem Protein führen zu kognitiven Beeinträchtigungen und Epilepsie, Zur Verfügung gestellt vom Autor
Die nächsten Schritte in diesem komplexen Bereich der Proteinwissenschaft bestehen darin, ihre Funktion zu entschlüsseln (d. h. zu verstehen, was sie tun, wenn sie interagieren), ihre Dynamik zu erforschen (d. h. wie sie während dieser Interaktionen ihre Form ändern) und zu untersuchen, wie unstrukturierte Proteine – z deren Struktur AlphaFold2 nicht vorhersagen kann und die etwa die Hälfte der menschlichen Proteine ausmachen – interagieren mit anderen Proteinen; und schließlich geht es darum, Proteine mit neuen Funktionen zu entwerfen, im Einklang mit den Fortschritten, die uns die synthetische Biologie in den kommenden Jahren bringen wird.
Diese vier großen Herausforderungen stehen im Mittelpunkt der aktuellen Forschung und ihre Lösung wird zu großen Durchbrüchen in der Biologie führen.
Zukünftig werden die beiden mit dem Nobelpreis für Chemie 2024 gewürdigten Fortschritte mit diesen neuen Fragestellungen verknüpft und so die Rekonstruktion von Interaktomen (Interaktionsnetzwerken zwischen Tausenden von Molekülen – einschließlich Proteinen – in einer Zelle oder einem biologischen Gewebe) im großen Stil ermöglichen Maßstab, ob völlig neu oder natürlich, für jede Art, jedes Individuum oder jede Gruppe von Organismen.